全面揭示猪肺部抗生素抗性基因的组成、宿主细菌、转移的可能路径及其与呼吸道病变的关系
2023年8月12日,买球赛的app官网猪遗传改良与种质创新全国重点实验室黄路生院士团队在国际重要学术刊物“自然子刊”Nature Communications在线发表题为“Characterization of the pig lower respiratory tract antibiotic resistome”(Doi: 10.1038/s41467-023-40587-1)的重要研究成果。该成果提供了首个猪下呼吸道微生物抗生素抗性基因(ARGs)图谱,描绘了猪下呼吸道耐药组特征,解析了抗生素抗性基因通过可移动遗传原件介导的在猪下呼吸道、肠道和人下呼吸道微生物之间的水平传播,本论文还首次研究了猪下呼吸道耐药组与肺部病变的关系。中国科学院院士、中国畜牧兽医学会理事长、买球赛的app官网猪遗传改良与种质创新全国重点实验室主任黄路生,买球赛的app官网首席教授陈从英为共同通讯作者,买球赛的app官网周云燕博士、李井泉博士、黄菲博士为共同第一作者。
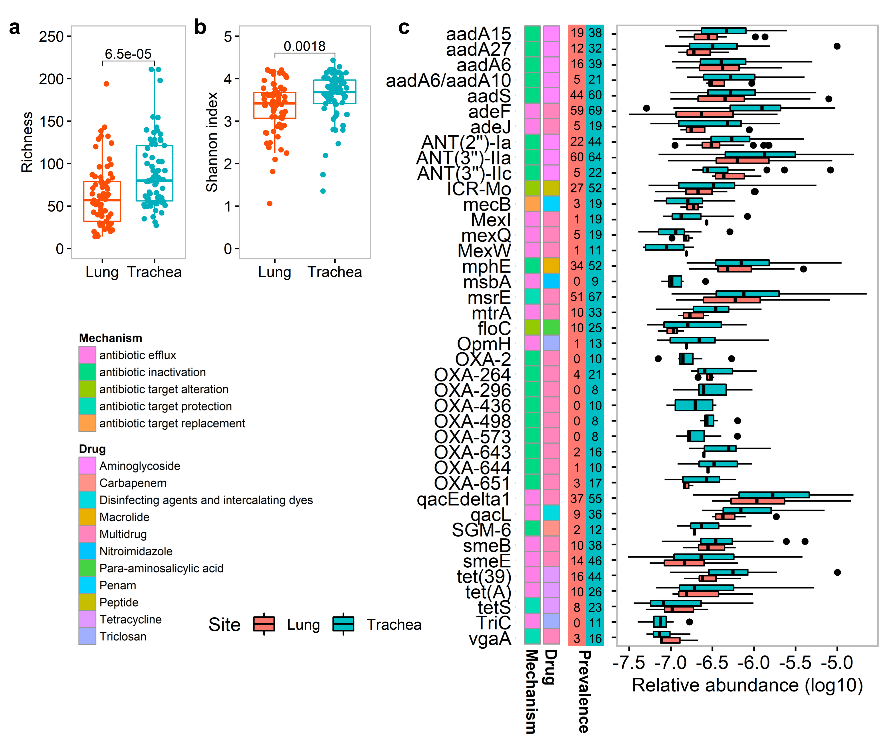
图:猪肺部和气管抗生素抗性基因组成比较
背景介绍:抗生素耐药性已成为全球人类健康的一大威胁。猪呼吸道疾病是养猪业面临的主要挑战之一,由于潜在的交叉感染风险,对人类健康也有重要影响。每年有大量抗生素用于猪呼吸道疾病的治疗,但目前猪的抗生素耐药性研究主要聚焦于肠道微生物,呼吸道微生物抗生素耐药组的特征及其与肺部健康的关系尚不清楚。了解猪呼吸道微生物组中抗生素抗性基因的组成、分布及其水平传播途径不仅可以指导猪呼吸道疾病治疗中抗生素的使用,对人类健康及生态环境保护也有重要意义。
主要创新:项目组用12年时间构建的由全世界四个不同商业品种及四个中国地方猪种混合杂交而成,且有精细肺部病变表型测定记录的嵌合家系第七世代群体为主要研究群体,结合实验室培育的获国家认定的“山下长黑”猪新品种,中国地方猪种二花脸、藏猪以及野猪等实验猪群体,测定了肺部和气管两个下呼吸道部位共计745个微生物样本,通过高深度宏基因组测序,取得了五个方面的创新性研究成果:
一、系统分析了猪下呼吸道微生物抗生素抗性基因的组成与分布,提供了首个猪下呼吸道微生物抗生素耐药组和可移动元件图谱。
从745个猪下呼吸道微生物样本中共鉴定到1228个编码抗生素抗性蛋白的开放阅读框(ORF),这些开放阅读框属于372种抗生素抗性基因类型和24种耐药类型。四环素类(33%)、氨基糖苷类(22%)抗性基因是猪下呼吸道最丰富的抗性基因类型。丰度最高的5种抗性基因是floR、tet(39)、tet(L)、tetQ和tet(D),在样本中分布最广的5种抗性基因是adeF、floR、tet(W/N/W)、APH(6)-Id和tetQ。几种重要的抗性基因,如sul1、Ermb和mcr在猪下呼吸道微生物组中存在较多的变体。
发现肺部微生物的抗性基因和可移动遗传元件的数量、总丰度和α多样性均显著高于气管,且β多样性也存在显著差异。共鉴别到40个抗性基因在肺部和气管微生物组中存在显著的丰度差异。不同猪种间抗性基因和可移动遗传元件的数量、总丰度和α多样性差异不显著,但β多样性存在显著差异。
二、发现了抗性基因与可移动遗传元件间的紧密联系,揭示了抗性基因在猪肺部和肠道微生物组以及人类肺部微生物组之间的潜在水平转移途径。
从抗性基因与可移动遗传元件间紧密联结(处于同一contig的 5 kb相邻范围内)以及共丰度两种方法鉴别到抗性基因与可移动遗传元件间的紧密联系,发现可移动遗传元件Tn916家族和四环素类抗性基因(尤其是tetM)、tnpA和多种不同类型抗性基因间紧密相关,提示Tn916家族和tnpA在相关抗性基因水平传播中起重要作用。利用课题组前期从猪肠道中组装得到的6339个微生物基因组数据(Nature Communications, 2021, 12: 1106. Doi:10.1038/s41467-021-21295-0)以及公共数据库中人类肺部微生物宏基因组测序数据,发现Tn916家族和tetM、tnpA和多种不同类型抗性基因间的紧密联系在猪肠道和人类肺部微生物中也存在。利用从人类和猪中分离得到的7种常见耐药菌的3878个基因组数据,发现几乎所有携带了tetM抗性基因的基因组同时也携带了Tn916家族基因。除tetM外,在Enterococcus faecalis 和Enterococcus faecium的部分菌株中还发现了Tn916家族与四环素抗性基因tet45、tet(W/N/W)间的紧密相关性。同时也验证了tnpA和不同类型抗性基因之间的紧密联系。这些结果提示Tn916家族和tnpA可在不同细菌种类中介导抗性基因在机体不同部位以及人类与猪之间的水平转移。
三、系统鉴别了抗生素抗性基因的宿主细菌,为肺部疾病治疗中抗生素使用提供参考。
研究发现γ变形菌纲(Gammaproteobacteria)是抗性基因的主要携带者,包含53%的抗性基因,其中大肠杆菌基因组上携带的抗性基因数量超过50个,可移动遗传元件数量超过 10个。有些抗性基因(如floR)的宿主细菌分布广泛甚至跨越不同的门。
四、首次揭示了肺部微生物抗生素抗性基因与肺部病变间的关系。
在613个精细测定了不同肺部病变程度(健康肺、轻度病变、中度病变和重度病变)个体中分析了抗生素抗性基因组成和丰度与肺部病变的关系,发现随着肺部病变程度增加,抗性基因组成的α多样性下降,进一步分析发现猪下呼吸道耐药组的组成主要由微生物群落的组成而不是肺部病变严重程度塑造。同时,还鉴别到73个抗性基因在不同病变程度猪肺中存在显著丰度差异。
五、深度宏基因组测序分析发现可移动遗传元件也能介导猪肺炎支原体毒力因子的水平转移,提示猪肺炎支原体感染风险。
研究发现猪肺炎支原体的平均丰度占肺部细菌总丰度的46%,且肺炎支原体丰度与猪肺部病变严重程度相关。然而,本研究组装得到以及公共数据库下载的共计30个猪肺炎支原体的基因组中均未鉴别到抗生素抗性基因,却发现其存在9个毒力因子,其中7个毒力因子具有细胞粘附功能,在这些毒力因子的临近区域同时还鉴定到可移动遗传元件的存在,包括前噬菌体、tnpA、IS91等,提示毒力因子可通过不同类型的移动遗传元件在菌株间水平转移,促进肺炎支原体的感染。
研究意义:本研究提供了首个猪肺部及气管微生物组的抗生素抗性基因集,揭示了猪肺部不同微生物携带的潜在可移动遗传元件介导的抗生素抗性基因在不同部位以及人类与猪之间的水平转移,全面鉴定了肺部抗生素抗性基因的宿主细菌,分析了抗性基因与肺部病变的关系。研究结果可为我国生猪生产中呼吸道疾病的治疗中抗生素的使用提供参考,对健康猪肉生产以及环境友好养殖保障具有重要意义。此外,人体肺部微生物样本采集困难,猪肺是人类肺部疾病研究良好的生物医学模型,且其微生物组成与人类肺部微生物组成具有一定的相似性,本研究构建的猪肺部及气管微生物组抗性基因图谱也可为人类下呼吸道微生物组抗性基因研究以及人类肺部疾病治疗中抗生素使用提供参考。
供稿:科技处 编发:宣传部

 赣公网安备 36010602000030
赣公网安备 36010602000030